ChemSpider、NCl Chemical Identifier Resolver
1.Pubchempy获取分子cid局限
import pubchempy as pcp
a = pcp.get_compounds('identifier','namespace')[0]
identifier填标识符,namespace填标识符类型
| 可选类型 |
|---|
| name |
| smiles |
| inchi |
| inchikey |
| formula |
在 PubChem 检索分子时,名称多来自文献或采购网站等渠道,质量参差不齐。为避免模糊匹配,直接使用
pcp.get_compounds(name, 'name')[0]
pubchempy可以完美避开模糊匹配情况,但是对于一些没有记载在数据库中的分子名,这种方法往往不能保持较高的效率。因此通过上述两种方法利用更广阔的名称检索分子smiles,保障准确性的同时提升pubchempy的成功匹配率。
2.ChemSpider🕷
ChemSpider的API文档
注册API教程
chemspider github网页
!pip install chemspipy
from chemspipy import ChemSpider
cs =ChemSpider('your_api_key')
search_name = cs.filter_name('compound_name')
results = cs.filter_results(search_name)
info = cs.get_details(results[0])
info['smiles']
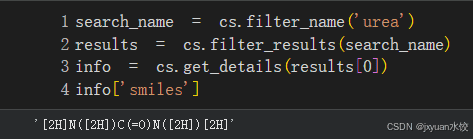
这样就可以找到smiles了,对于无法在Pubchem中搜索到的分子同样有效(部分):
举例:cellulose acetate(醋酸纤维素)
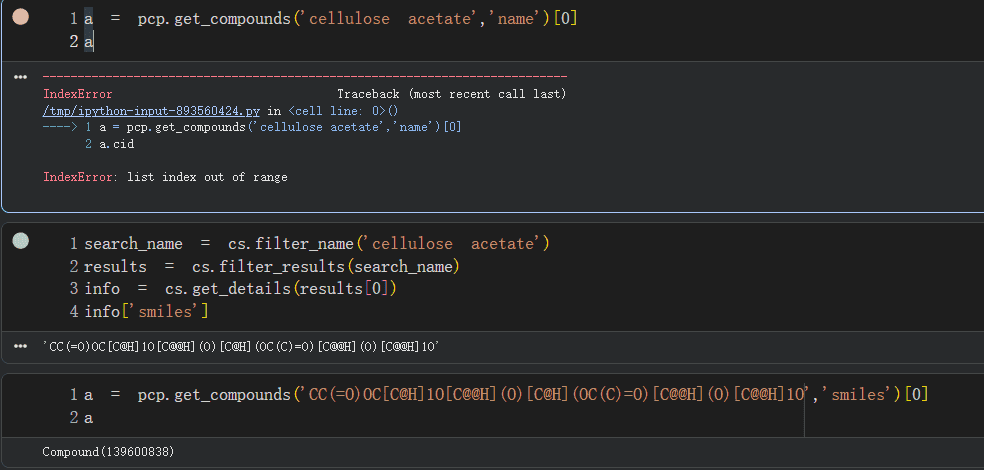
当使用pubchempy使用名字匹配分子时,由于改分子名称没有在Pubchempy的分子名称记录中,因此返回空列表,那么空列表的零号元素会直接报错,因此pubchem并没有叫这个名字的分子。
但是如果使用ChemSpider的filter_name功能,则能直接匹配到分子,这是因为不同数据库对分子名称的收集程度不同,显然就这个分子而言,ChemSpider涵盖了Pubchem没有的分子名称,所以他会返回一个smiles,当你想获得Pubchempy返回的全面分子属性时,可以再用这个smiles去pubchem匹配cid最终返回分子属性,如第三个单元格所示,pubchem成功使用smiles返回cid
为了验证ChemSpider提供的信息以及pubchem使用smiles匹配的cid是否正确,可以去ChemicalBook等卖分子的网站去寻找醋酸纤维素的分子结构,并且与pubchem搜寻到的进行对比:
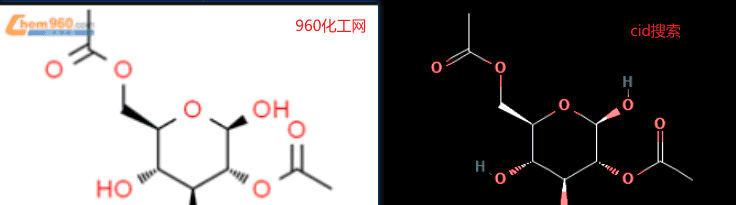
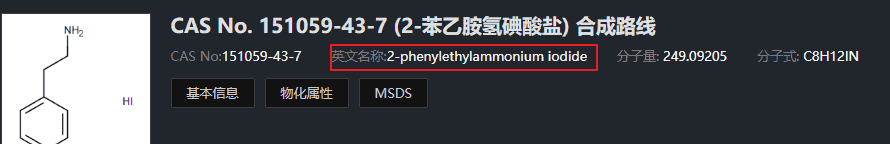
再比如:2-phenylethylammonium iodide
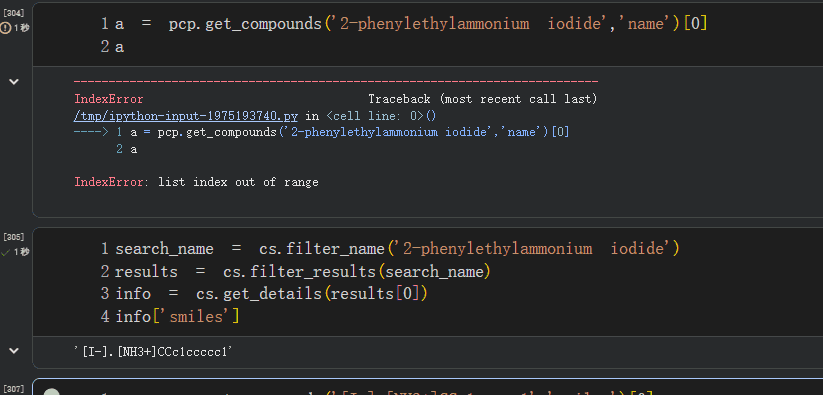
同样利用cid去pubchem搜索结构:
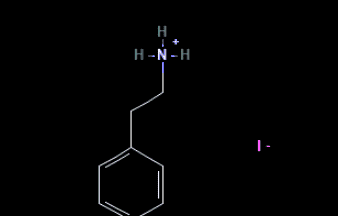
通过这两个例子可以看到ChemSpider的能力,它可以凭借有时更丰富的分子名来弥补Pubchempy的name搜索功能,可以帮助我们利用pubchempy尽可能多的获得信息,一般可以在pubchempy批量作业之后,在利用ChemSpider扫扫残余。
3.NCI Chemical Identifier Resolver
这是NCI的网址
NCI是一个功能强大的网站,它能将来自不同资料的分子描述符号转化你想要的格式,比如name转smiles,name转stdinchikey,转GIF Image,转ChemSpider ID,转 Chemical Formula等等。同时提供url格式访问,具体见上面的网址,下面是官网提供的url:
https://cactus.nci.nih.gov/chemical/structure/“structure identifier”/“representation”
在structure identifier处可以换成在规定范围内的任意描述符,转化成representation形式,(在规定范围内的任意描述符)
举例:pubchem无法识别的name标签转为smiles
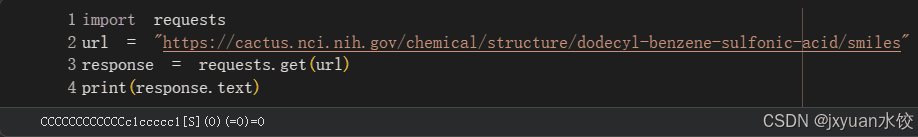
import requests
url = https://cactus.nci.nih.gov/chemical/structure/dodecyl-benzene-sulfonic-acid/smiles
response = requests.get(url)
print(response.text)
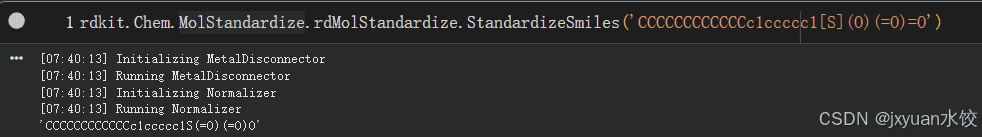
使用rdkit.Chem.MolStandardize.rdMolStandardize.StandardizeSmiles标准化smiles字符串
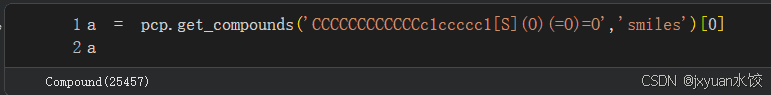
之后利用pubchem读取该smiles:
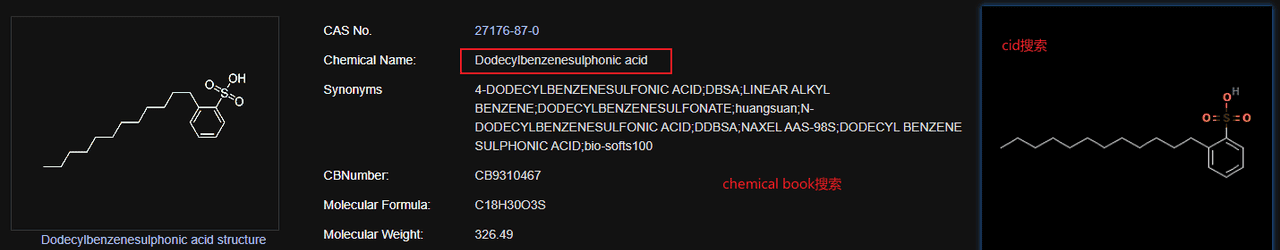
例子2:聚丙聚腈
相关文章