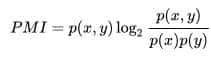前言:生信二次开发的价值与痛点
随着高通量测序技术的普及,生信分析从 “标准化流程” 走向 “个性化需求” 已成必然趋势。然而,现有开源工具(如 FastQC、DESeq2、Seurat)虽覆盖了基础分析场景,却难以直接满足特定研究需求 —— 例如肿瘤单细胞数据的自定义细胞分型、罕见病外显子数据的致病性变异筛选、宏基因组样本的特异性代谢通路富集等。
这种 “通用工具” 与 “专属需求” 的矛盾,催生了生信工具二次开发的需求。但开发者常面临三大痛点:
技术割裂:Python 生态(Pandas、Scikit-learn)与 R 生态(tidyverse、Bioconductor)工具链不互通,跨语言调用效率低;模块耦合:直接修改开源工具源码易导致 “牵一发而动全身”,后续难以同步上游更新;规范缺失:个人开发的模块无统一文档、测试用例和版本管理,无法融入团队协作或开源社区。
本文以 “封装个性化分析模块” 为核心,通过 Python 和 R 双语言实战案例,结合 GitHub 开源规范,提供一套 “低耦合、高复用、可共享” 的二次开发方案,帮助生信研究者将自定义分析逻辑转化为标准化工具。
第一章 开发前准备:环境搭建与需求拆解
在动手编码前,需完成 “工具选型 – 环境配置 – 需求定义” 三步准备,避免后续开发中频繁返工。
1.1 技术栈选型:Python 与 R 的场景适配
Python 和 R 是生信分析的两大主流语言,二者各有侧重,需根据需求选择核心开发语言:
| 维度 | Python 生态优势 | R 生态优势 | 生信场景适配 |
|---|---|---|---|
| 数据处理 | 大样本(10G+)数据高效读写(Pandas) | 小样本精细化清洗(dplyr、tidyr) | 单细胞矩阵(Python)、临床表型(R) |
| 可视化 | 交互式图表(Plotly)、批量出图 | 统计图形(ggplot2)、生物信息专用图(pheatmap) | 动态报告(Python)、论文级静态图(R) |
| 生物信息库 | PyTorch(深度学习)、Scanpy(单细胞) | Bioconductor(1900 + 包)、DESeq2(差异分析) | AI 辅助分析(Python)、经典统计流程(R) |
| 跨语言调用 | 通过 |
通过 |
混合开发时优先选 “核心逻辑 + 辅助调用” 模式 |
实战建议:若核心需求是 “差异表达分析后自定义功能富集”,优先用 R 作为开发语言(直接复用 DESeq2 结果);若需 “单细胞数据降维后机器学习聚类”,选择 Python(依托 Scanpy+Scikit-learn 生态)。
1.2 开发环境配置(Windows/macOS/Linux 通用)
1.2.1 Python 环境(以 Anaconda 为例)
bash
# 1. 创建专用虚拟环境(避免依赖冲突)
conda create -n bio_dev python=3.9 -y
conda activate bio_dev
# 2. 安装核心依赖
conda install pandas numpy matplotlib seaborn -y # 基础数据处理与可视化
conda install scanpy scikit-learn -y # 单细胞分析与机器学习
conda install rpy2 -y # 调用 R 包(若需跨语言)
conda install pytest sphinx -y # 测试与文档生成1.2.2 R 环境(以 RStudio 为例)
r
# 1. 安装 Bioconductor 管理工具(生信包核心源)
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
# 2. 安装核心依赖
BiocManager::install(c("tidyverse", "DESeq2", "SummarizedExperiment")) # 基础与差异分析
BiocManager::install(c("pheatmap", "ggplot2", "patchwork")) # 可视化
BiocManager::install(c("reticulate", "testthat", "devtools")) # 跨语言调用与开发工具1.3 需求拆解:从 “模糊需求” 到 “可编码功能”
以 “肿瘤单细胞 RNA 测序数据的自定义免疫细胞分型模块” 为例,演示需求拆解流程:
明确输入输出
输入:Scanpy 格式的
AnnData
AnnData
拆解核心功能
功能 1:特征基因导入与验证(检查输入基因是否在表达矩阵中存在);功能 2:免疫细胞评分计算(基于特征基因的平均表达量或 AUC 值);功能 3:基于评分的细胞分型(设定阈值或聚类算法划分细胞类型);功能 4:结果可视化(UMAP 图标注分型、特征基因热图);功能 5:异常值处理(过滤低质量细胞、缺失值填充)。
定义接口规范提前约定函数 / 模块的调用方式,避免后续开发中接口频繁变更。例如 Python 模块的核心函数接口:
python
def custom_immune_typing(
adata: anndata.AnnData, # 输入AnnData对象
marker_gene_path: str, # 特征基因列表路径
scoring_method: str = "mean", # 评分方法(mean/AUC)
threshold: float = 0.5, # 分型阈值
plot_save_path: str = "./plots" # 可视化结果保存路径
) -> tuple[anndata.AnnData, pd.DataFrame]:
"""
肿瘤单细胞数据的自定义免疫细胞分型
Returns:
更新后的AnnData对象、分型统计表格
"""
pass第二章 Python 实战:封装个性化分析模块
以 “单细胞免疫细胞分型” 为例,完整演示 Python 模块的封装流程,涵盖 “核心逻辑实现 – 参数校验 – 可视化集成 – 跨语言调用”。
2.1 模块结构设计(低耦合原则)
采用 “功能拆分 + 分层调用” 的结构,避免将所有代码堆在一个脚本中。标准目录如下:
plaintext
custom_immune_typing/ # 模块根目录
├── __init__.py # 模块入口(暴露核心函数)
├── core.py # 核心逻辑(评分计算、分型)
├── io.py # 输入输出(基因列表读取、结果保存)
├── plot.py # 可视化(UMAP图、热图)
└── utils.py # 工具函数(异常值处理、基因验证)2.2 核心代码实现(逐文件讲解)
2.2.1 utils.py(工具函数:基因验证与异常值处理)
python
import anndata as ad
import pandas as pd
from typing import List
def validate_marker_genes(adata: ad.AnnData, marker_genes: List[str]) -> List[str]:
"""
验证特征基因是否在AnnData对象中存在,返回存在的基因列表
"""
# 获取表达矩阵中的基因名
adata_genes = adata.var_names.tolist()
# 筛选存在的基因
valid_genes = [gene for gene in marker_genes if gene in adata_genes]
# 提示缺失的基因
missing_genes = set(marker_genes) - set(valid_genes)
if missing_genes:
print(f"警告:以下基因在表达矩阵中缺失,已自动过滤:{missing_genes}")
# 若有效基因为空,抛出异常
if not valid_genes:
raise ValueError("所有特征基因均不在表达矩阵中,请检查基因列表")
return valid_genes
def filter_low_quality_cells(adata: ad.AnnData, min_genes: int = 200) -> ad.AnnData:
"""
过滤低质量细胞(基因检测数小于min_genes的细胞)
"""
before_filter = adata.n_obs
adata = adata[adata.obs["n_genes"] >= min_genes, :]
after_filter = adata.n_obs
print(f"过滤低质量细胞:{before_filter} → {after_filter}(保留{after_filter/before_filter:.2%})")
return adata2.2.2 io.py(输入输出:基因列表读取与结果保存)
python
import pandas as pd
import os
from typing import Dict
def read_marker_genes(marker_gene_path: str) -> Dict[str, List[str]]:
"""
读取特征基因列表(CSV格式,列:cell_type, marker_genes)
返回:字典{细胞类型: [基因1, 基因2, ...]}
"""
if not os.path.exists(marker_gene_path):
raise FileNotFoundError(f"特征基因文件不存在:{marker_gene_path}")
# 读取CSV并处理(基因列表用逗号分隔)
marker_df = pd.read_csv(marker_gene_path)
if not {"cell_type", "marker_genes"}.issubset(marker_df.columns):
raise ValueError("特征基因文件需包含'cell_type'和'marker_genes'列")
# 转换为字典
marker_dict = {}
for _, row in marker_df.iterrows():
cell_type = row["cell_type"]
genes = [gene.strip() for gene in row["marker_genes"].split(",")]
marker_dict[cell_type] = genes
return marker_dict
def save_typing_result(stat_df: pd.DataFrame, save_path: str = "./results"):
"""
保存分型统计结果
"""
os.makedirs(save_path, exist_ok=True)
save_file = os.path.join(save_path, "immune_typing_stat.csv")
stat_df.to_csv(save_file, index=False)
print(f"分型统计结果已保存至:{save_file}")2.2.3 core.py(核心逻辑:评分计算与细胞分型)
python
import anndata as ad
import pandas as pd
import numpy as np
from sklearn.metrics import roc_auc_score
from .utils import validate_marker_genes
from .io import read_marker_genes
def calculate_gene_score(adata: ad.AnnData, marker_genes: List[str], method: str = "mean") -> np.ndarray:
"""
计算细胞的特征基因评分
method: mean(平均表达量)、AUC(基因表达是否高于背景)
"""
# 验证基因并提取表达矩阵
valid_genes = validate_marker_genes(adata, marker_genes)
expr_matrix = adata[:, valid_genes].X # (n_cells, n_genes)
# 计算评分
if method == "mean":
# 平均表达量(排除0值)
expr_matrix[expr_matrix < 0] = 0 # 处理可能的负数值(如标准化后)
score = np.mean(expr_matrix, axis=1)
elif method == "AUC":
# AUC评分(将每个细胞的基因表达与背景比较)
background = np.random.normal(
loc=np.mean(adata.X),
scale=np.std(adata.X),
size=expr_matrix.shape
)
score = np.array([roc_auc_score(expr, bg) for expr, bg in zip(expr_matrix, background)])
else:
raise ValueError("评分方法仅支持'mean'或'AUC'")
return score
def assign_cell_type(
adata: ad.AnnData,
marker_gene_path: str,
scoring_method: str = "mean",
threshold: float = 0.5
) -> tuple[ad.AnnData, pd.DataFrame]:
"""
为细胞分配类型,返回更新后的AnnData和统计表格
"""
# 1. 读取特征基因
marker_dict = read_marker_genes(marker_gene_path)
# 2. 计算每种细胞类型的评分
score_df = pd.DataFrame(index=adata.obs_names)
for cell_type, genes in marker_dict.items():
score_df[cell_type] = calculate_gene_score(adata, genes, scoring_method)
# 3. 分配细胞类型(取最高评分且超过阈值)
max_score = score_df.max(axis=1)
max_type = score_df.idxmax(axis=1)
adata.obs["custom_immune_type"] = np.where(max_score >= threshold, max_type, "Unknown")
# 4. 生成统计表格
stat_df = adata.obs["custom_immune_type"].value_counts().reset_index()
stat_df.columns = ["cell_type", "cell_count"]
stat_df["percentage"] = stat_df["cell_count"] / stat_df["cell_count"].sum() * 100
return adata, stat_df2.2.4 plot.py(可视化:结果展示)
python
import anndata as ad
import matplotlib.pyplot as plt
import seaborn as sns
import os
from typing import List
def plot_umap_typing(adata: ad.AnnData, save_path: str = "./plots"):
"""
绘制UMAP图,标注自定义细胞分型
"""
os.makedirs(save_path, exist_ok=True)
plt.figure(figsize=(10, 8))
# 检查是否有UMAP坐标(若没有则先计算)
if "X_umap" not in adata.obsm.keys():
print("UMAP坐标不存在,将自动计算...")
import scanpy as sc
sc.tl.umap(adata)
# 绘制UMAP
sns.scatterplot(
x=adata.obsm["X_umap"][:, 0],
y=adata.obsm["X_umap"][:, 1],
hue=adata.obs["custom_immune_type"],
s=10, # 点大小
palette="tab20", # 颜色板
legend="full"
)
plt.title("Custom Immune Cell Typing (UMAP)", fontsize=14)
plt.legend(bbox_to_anchor=(1.05, 1), loc="upper left")
plt.tight_layout()
plt.savefig(os.path.join(save_path, "umap_typing.png"), dpi=300)
plt.close()
print(f"UMAP分型图已保存至:{save_path}/umap_typing.png")
def plot_marker_heatmap(adata: ad.AnnData, marker_genes: List[str], save_path: str = "./plots"):
"""
绘制特征基因热图(按细胞类型分组)
"""
os.makedirs(save_path, exist_ok=True)
# 筛选细胞类型(排除Unknown)
adata_filtered = adata[adata.obs["custom_immune_type"] != "Unknown", :]
if adata_filtered.n_obs == 0:
raise ValueError("无有效细胞类型可绘制热图")
# 提取特征基因表达矩阵
valid_genes = [gene for gene in marker_genes if gene in adata_filtered.var_names]
expr_matrix = adata_filtered[:, valid_genes].X.T # (n_genes, n_cells)
# 按细胞类型排序
cell_order = adata_filtered.obs.sort_values("custom_immune_type").index
expr_matrix = expr_matrix[:, [adata_filtered.obs_names.get_loc(cell) for cell in cell_order]]
# 绘制热图
plt.figure(figsize=(12, 8))
sns.heatmap(
expr_matrix,
cmap="viridis",
yticklabels=valid_genes,
xticklabels=False, # 细胞过多时隐藏x轴标签
cbar_kws={"label": "Normalized Expression"}
)
# 添加细胞类型分隔线
cell_type_counts = adata_filtered.obs["custom_immune_type"].value_counts().sort_index()
cumulative_counts = cell_type_counts.cumsum()[:-1]
for count in cumulative_counts:
plt.axvline(x=count, color="white", linewidth=2)
plt.title("Marker Genes Expression Heatmap", fontsize=14)
plt.tight_layout()
plt.savefig(os.path.join(save_path, "marker_heatmap.png"), dpi=300)
plt.close()
print(f"特征基因热图已保存至:{save_path}/marker_heatmap.png")2.2.5 init.py(模块入口:暴露核心函数)
python
"""
自定义免疫细胞分型模块(Python)
用于肿瘤单细胞RNA测序数据的个性化细胞注释
"""
# 暴露核心函数(方便用户调用)
from .core import assign_cell_type
from .plot import plot_umap_typing, plot_marker_heatmap
from .io import save_typing_result
from .utils import filter_low_quality_cells
# 模块版本(遵循语义化版本:主版本.次版本.修订号)
__version__ = "0.1.0"
# 作者与联系方式(便于协作)
__author__ = "Bioinformatics Developer"
__email__ = "dev@bioinfo.example.com"2.3 模块测试:确保功能稳定性
采用
pytest
2.3.1 测试用例编写(test_custom_immune.py)
python
import anndata as ad
import pandas as pd
import numpy as np
import os
import tempfile
from custom_immune_typing import (
assign_cell_type,
filter_low_quality_cells,
read_marker_genes
)
# 1. 生成测试用的AnnData对象
def create_test_adata() -> ad.AnnData:
n_cells = 100
n_genes = 50
# 模拟表达矩阵(泊松分布,模拟单细胞数据)
expr_matrix = np.random.poisson(lam=0.5, size=(n_cells, n_genes))
# 模拟基因名(Gene1-Gene50)
var_names = [f"Gene{i}" for i in range(1, n_genes+1)]
# 模拟细胞名(Cell1-Cell100)
obs_names = [f"Cell{i}" for i in range(1, n_cells+1)]
# 模拟n_genes(用于低质量细胞过滤)
obs = pd.DataFrame(
{"n_genes": np.random.randint(100, 500, size=n_cells)},
index=obs_names
)
# 创建AnnData对象
adata = ad.AnnData(
X=expr_matrix,
obs=obs,
var=pd.DataFrame(index=var_names)
)
return adata
# 2. 生成测试用的特征基因列表
def create_test_marker_genes() -> str:
# 创建临时CSV文件
marker_df = pd.DataFrame({
"cell_type": ["T_cell", "B_cell", "Macrophage"],
"marker_genes": ["Gene1,Gene2,Gene3", "Gene4,Gene5,Gene6", "Gene7,Gene8,Gene9"]
})
temp_file = tempfile.NamedTemporaryFile(mode="w", suffix=".csv", delete=False)
marker_df.to_csv(temp_file, index=False)
temp_file.close()
return temp_file.name
# 3. 测试函数
def test_filter_low_quality_cells():
adata = create_test_adata()
# 过滤低质量细胞(min_genes=300)
adata_filtered = filter_low_quality_cells(adata, min_genes=300)
# 验证过滤结果(n_genes>=300的细胞保留)
assert all(adata_filtered.obs["n_genes"] >= 300)
assert adata_filtered.n_obs <= adata.n_obs
def test_read_marker_genes():
marker_path = create_test_marker_genes()
marker_dict = read_marker_genes(marker_path)
# 验证字典结构
assert set(marker_dict.keys()) == {"T_cell", "B_cell", "Macrophage"}
assert len(marker_dict["T_cell"]) == 3
# 删除临时文件
os.unlink(marker_path)
def test_assign_cell_type():
adata = create_test_adata()
marker_path = create_test_marker_genes()
# 分配细胞类型
adata_typed, stat_df = assign_cell_type(adata, marker_path, threshold=0.1)
# 验证结果
assert "custom_immune_type" in adata_typed.obs.columns
assert set(stat_df["cell_type"]) >= {"T_cell", "B_cell", "Macrophage", "Unknown"}
assert np.isclose(stat_df["percentage"].sum(), 100, atol=0.1)
# 删除临时文件
os.unlink(marker_path)2.3.2 执行测试
bash
# 在模块根目录执行
pytest test_custom_immune.py -v若所有测试用例通过(显示
PASSED
FAILED
第三章 R 实战:封装个性化分析模块
以 “差异表达基因的自定义功能富集分析” 为例,演示 R 模块的封装流程,重点覆盖 “Bioconductor 包复用”“S3 类定义”“文档生成”。
3.1 模块结构设计(符合 R 包规范)
R 模块通常以 “包” 的形式封装,便于安装和调用,标准目录如下:
plaintext
CustomEnrichment/ # 包根目录
├── DESCRIPTION # 包描述(名称、版本、依赖)
├── NAMESPACE # 命名空间(导出函数)
├── R/ # 核心代码目录
│ ├── core_functions.R # 富集分析核心逻辑
│ ├── io_functions.R # 输入输出(读取GMT文件、保存结果)
│ └── plot_functions.R # 可视化(富集气泡图、条形图)
├── man/ # 帮助文档(自动生成)
└── tests/ # 测试用例
└── testthat/
└── test-core.R # 核心函数测试3.2 核心文件编写
3.2.1 DESCRIPTION(包描述文件)
plaintext
Package: CustomEnrichment
Type: Package
Title: Custom Functional Enrichment for Differentially Expressed Genes
Version: 0.1.0
Authors@R: person("Bioinformatics", "Developer", email = "dev@bioinfo.example.com", role = c("aut", "cre"))
Description: A tool for custom functional enrichment analysis of differentially expressed genes (DEGs) from RNA-seq data, supporting user-defined gene sets and multiple enrichment statistics.
License: MIT + file LICENSE
Imports:
tidyverse,
clusterProfiler,
enrichplot,
org.Hs.eg.db, # 人类基因注释库(根据物种选择)
testthat (>= 3.0.0)
Suggests:
knitr,
rmarkdown
Encoding: UTF-8
LazyData: true
RoxygenNote: 7.2.33.2.2 R/core_functions.R(核心逻辑:富集分析)
r
#' 自定义功能富集分析
#'
#' 基于用户提供的基因集,对差异表达基因进行富集分析,支持超几何检验和Fisher精确检验
#'
#' @param de_genes 字符向量,差异表达基因列表(需与基因集中的基因名匹配)
#' @param background_genes 字符向量,背景基因列表(通常为所有检测到的基因)
#' @param gene_set 数据框,用户定义的基因集,包含两列:term(功能术语)、gene(基因名)
#' @param test_method 字符,检验方法,可选"hypergeometric"(超几何检验)或"fisher"(Fisher精确检验)
#' @param pvalue_cutoff 数值,P值阈值,默认0.05
#' @param qvalue_cutoff 数值,Q值阈值(FDR校正后),默认0.1
#'
#' @return 列表,包含富集结果(data.frame)和分析参数(list)
#' @export
#' @examples
#' # 1. 准备数据
#' de_genes <- c("TP53", "BRCA1", "EGFR")
#' background_genes <- c("TP53", "BRCA1", "EGFR", "AKT1", "MYC", "VEGFA")
#' gene_set <- data.frame(
#' term = rep(c("Apoptosis", "DNA Repair"), each = 3),
#' gene = c("TP53", "BRCA1", "BAX", "BRCA1", "ATM", "EGFR")
#' )
#' # 2. 执行富集分析
#' result <- custom_enrich(
#' de_genes = de_genes,
#' background_genes = background_genes,
#' gene_set = gene_set,
#' test_method = "hypergeometric"
#' )
#' # 3. 查看结果
#' head(result$enrichment_result)
custom_enrich <- function(
de_genes,
background_genes,
gene_set,
test_method = c("hypergeometric", "fisher"),
pvalue_cutoff = 0.05,
qvalue_cutoff = 0.1
) {
# 1. 参数校验
test_method <- match.arg(test_method)
if (!all(c("term", "gene") %in% colnames(gene_set))) {
stop("基因集必须包含'term'和'gene'列")
}
if (length(de_genes) == 0) {
stop("差异表达基因列表不能为空")
}
# 过滤背景基因中不存在的DE基因
de_genes <- intersect(de_genes, background_genes)
if (length(de_genes) == 0) {
stop("所有差异表达基因均不在背景基因列表中,请检查基因名匹配")
}
# 2. 整理基因集(按term分组)
gene_set_list <- gene_set %>%
dplyr::group_by(term) %>%
dplyr::summarise(gene = list(unique(gene)), .groups = "drop") %>%
tibble::deframe() # 转换为列表:term -> 基因向量
# 3. 执行富集检验
enrichment_result <- switch(
test_method,
"hypergeometric" = run_hypergeometric_test(de_genes, background_genes, gene_set_list),
"fisher" = run_fisher_test(de_genes, background_genes, gene_set_list)
)
# 4. 过滤显著结果(P值和Q值)
enrichment_result <- enrichment_result %>%
dplyr::mutate(qvalue = p.adjust(pvalue, method = "fdr")) %>%
dplyr::filter(pvalue <= pvalue_cutoff & qvalue <= qvalue_cutoff) %>%
dplyr::arrange(pvalue) # 按P值排序
# 5. 返回结果(包含参数信息)
result <- list(
enrichment_result = enrichment_result,
parameters = list(
test_method = test_method,
pvalue_cutoff = pvalue_cutoff,
qvalue_cutoff = qvalue_cutoff,
de_gene_count = length(de_genes),
background_gene_count = length(background_genes)
)
)
# 为结果添加S3类(便于后续可视化函数识别)
class(result) <- "CustomEnrichResult"
return(result)
}
#' 超几何检验(内部函数,不导出)
run_hypergeometric_test <- function(de_genes, background_genes, gene_set_list) {
# 超几何分布参数:x=成功次数,m=总体中的成功次数,n=总体中的失败次数,k=抽取次数
# x:基因集中与DE基因重叠的数量
# m:背景基因中DE基因的总数
# n:背景基因中非DE基因的总数
# k:基因集中的基因总数
m <- length(de_genes)
n <- length(background_genes) - m
# 对每个term计算P值
term_stats <- purrr::map_dfr(names(gene_set_list), function(term) {
gene_set_genes <- gene_set_list[[term]]
k <- length(gene_set_genes)
# 计算重叠基因数
x <- length(intersect(gene_set_genes, de_genes))
# 超几何检验(下侧检验,P值=P(X>=x))
pvalue <- 1 - phyper(x - 1, m, n, k)
# 返回统计信息
tibble::tibble(
term = term,
gene_set_size = k,
overlap_gene_count = x,
overlap_genes = paste(intersect(gene_set_genes, de_genes), collapse = ","),
pvalue = pvalue
)
})
return(term_stats)
}
#' Fisher精确检验(内部函数,不导出)
run_fisher_test <- function(de_genes, background_genes, gene_set_list) {
# 2x2列联表:
# 基因集内 基因集外
# DE基因 a b
# 非DE基因 c d
# 对每个term计算P值
term_stats <- purrr::map_dfr(names(gene_set_list), function(term) {
gene_set_genes <- gene_set_list[[term]]
# 计算列联表数值
a <- length(intersect(gene_set_genes, de_genes))
b <- length(setdiff(de_genes, gene_set_genes))
c <- length(setdiff(gene_set_genes, de_genes))
d <- length(setdiff(background_genes, union(gene_set_genes, de_genes)))
# 构建列联表
contingency_table <- matrix(c(a, c, b, d), nrow = 2, byrow = TRUE)
# Fisher精确检验(双侧检验)
fisher_result <- fisher.test(contingency_table, alternative = "two.sided")
# 返回统计信息
tibble::tibble(
term = term,
gene_set_size = a + c,
overlap_gene_count = a,
overlap_genes = paste(intersect(gene_set_genes, de_genes), collapse = ","),
pvalue = fisher_result$p.value
)
})
return(term_stats)
}3.2.3 R/plot_functions.R(可视化:富集结果展示)
r
#' 绘制富集结果气泡图
#'
#' 为CustomEnrichResult类对象绘制气泡图,展示富集术语的显著性、基因集大小和重叠基因数
#'
#' @param x CustomEnrichResult类对象,由custom_enrich()生成
#' @param n_terms 数值,展示的术语数量,默认15
#' @param save_path 字符,图片保存路径(如"./enrich_bubble.png"),默认NULL(不保存)
#' @param ... 其他参数(如width、height,传递给ggsave())
#'
#' @return ggplot2对象
#' @export
#' @method plot CustomEnrichResult
plot.CustomEnrichResult <- function(x, n_terms = 15, save_path = NULL, ...) {
# 提取富集结果
enrich_df <- x$enrichment_result
if (nrow(enrich_df) == 0) {
stop("无显著富集的术语,无法绘制气泡图")
}
# 筛选前n_terms个术语(按P值排序)
plot_df <- enrich_df %>%
dplyr::slice_head(n = n_terms) %>%
dplyr::mutate(term = factor(term, levels = rev(term))) # 倒序排列,便于查看
# 绘制气泡图
p <- ggplot2::ggplot(plot_df, ggplot2::aes(x = -log10(pvalue), y = term)) +
ggplot2::geom_point(
ggplot2::aes(size = overlap_gene_count, color = qvalue),
alpha = 0.7
) +
# 颜色配置(qvalue越小,颜色越深)
ggplot2::scale_color_gradient(low = "red", high = "blue") +
# 大小配置(重叠基因数越多,点越大)
ggplot2::scale_size(range = c(3, 10)) +
# 标签与主题
ggplot2::labs(
x = "-log10(P-value)",
y = "Functional Term",
color = "Q-value (FDR)",
size = "Overlap Gene Count",
title = "Custom Functional Enrichment Result"
) +
ggplot2::theme_bw() +
ggplot2::theme(
plot.title = ggplot2::element_text(hjust = 0.5, size = 14),
axis.text.y = ggplot2::element_text(size = 10)
)
# 保存图片(若指定路径)
if (!is.null(save_path)) {
ggplot2::ggsave(save_path, plot = p, ...)
message(paste("气泡图已保存至:", save_path))
}
return(p)
}
#' 绘制富集结果条形图
#'
#' 展示每个术语的重叠基因数和P值
#'
#' @param enrich_result CustomEnrichResult类对象
#' @param n_terms 数值,展示的术语数量,默认10
#' @param save_path 字符,图片保存路径,默认NULL
#'
#' @return ggplot2对象
#' @export
plot_enrich_bar <- function(enrich_result, n_terms = 10, save_path = NULL) {
# 校验输入类型
if (!inherits(enrich_result, "CustomEnrichResult")) {
stop("输入必须是CustomEnrichResult类对象,请先运行custom_enrich()")
}
enrich_df <- enrich_result$enrichment_result
if (nrow(enrich_df) == 0) {
stop("无显著富集的术语,无法绘制条形图")
}
# 筛选术语并处理
plot_df <- enrich_df %>%
dplyr::slice_head(n = n_terms) %>%
dplyr::mutate(
term = factor(term, levels = term[order(pvalue)]), # 按P值排序
pvalue_label = paste0("P=", round(pvalue, 4))
)
# 绘制条形图
p <- ggplot2::ggplot(plot_df, ggplot2::aes(x = term, y = overlap_gene_count)) +
ggplot2::geom_bar(stat = "identity", fill = "steelblue", alpha = 0.7) +
ggplot2::geom_text(
ggplot2::aes(label = pvalue_label),
vjust = -0.3, size = 3.5
) +
ggplot2::coord_flip() +
ggplot2::labs(
x = "Functional Term",
y = "Overlap Gene Count",
title = "Custom Enrichment: Overlap Genes per Term"
) +
ggplot2::theme_bw() +
ggplot2::theme(
plot.title = ggplot2::element_text(hjust = 0.5, size = 14),
axis.text.x = ggplot2::element_text(size = 10)
)
# 保存图片
if (!is.null(save_path)) {
ggplot2::ggsave(save_path, plot = p, width = 10, height = 6, dpi = 300)
message(paste("条形图已保存至:", save_path))
}
return(p)
}3.2.4 R/io_functions.R(输入输出:基因集读取与结果保存)
r
#' 读取GMT格式的基因集文件
#'
#' GMT格式(Gene Matrix Transposed)是生信中常用的基因集格式,每行格式:term<TAB>description<TAB>gene1<TAB>gene2<TAB>...
#'
#' @param gmt_path 字符,GMT文件路径
#'
#' @return 数据框,包含term和gene两列
#' @export
read_gmt <- function(gmt_path) {
if (!file.exists(gmt_path)) {
stop(paste("GMT文件不存在:", gmt_path))
}
# 读取GMT文件(每行分割为term、description、基因列表)
gmt_lines <- readLines(gmt_path)
gene_set_df <- purrr::map_dfr(gmt_lines, function(line) {
parts <- strsplit(line, " ")[[1]]
term <- parts[1]
# 跳过description(parts[2]),提取基因
genes <- parts[-c(1, 2)]
# 过滤空基因
genes <- genes[genes != ""]
if (length(genes) == 0) {
warning(paste("术语", term, "无基因,已跳过"))
return(NULL)
}
# 返回数据框行
tibble::tibble(term = term, gene = genes)
})
return(gene_set_df)
}
#' 保存富集分析结果
#'
#' 将富集结果保存为CSV文件,包含术语、P值、Q值、重叠基因等信息
#'
#' @param enrich_result CustomEnrichResult类对象
#' @param save_path 字符,保存路径(如"./enrich_result.csv")
#'
#' @export
save_enrich_result <- function(enrich_result, save_path) {
if (!inherits(enrich_result, "CustomEnrichResult")) {
stop("输入必须是CustomEnrichResult类对象")
}
# 提取结果并添加参数信息
result_df <- enrich_result$enrichment_result
param_df <- tibble::tibble(
parameter = names(enrich_result$parameters),
value = as.character(enrich_result$parameters)
)
# 保存为CSV(前几行是参数,后面是结果)
writeLines("## Enrichment Analysis Parameters", con = save_path)
readr::write_csv(param_df, file = save_path, append = TRUE)
writeLines("
## Enrichment Result", con = save_path, append = TRUE)
readr::write_csv(result_df, file = save_path, append = TRUE)
message(paste("富集结果已保存至:", save_path))
}3.2.5 NAMESPACE(命名空间:导出函数)
通过
roxygen2
@export
devtools::document()
3.3 文档生成与包安装
3.3.1 生成帮助文档
r
# 在RStudio中执行(需安装devtools和roxygen2)
devtools::document()执行后会在
man/
custom_enrich.Rd
?custom_enrich
3.3.2 安装包并测试
r
# 1. 安装包(从本地目录)
devtools::install_local("path/to/CustomEnrichment", dependencies = TRUE)
# 2. 加载包
library(CustomEnrichment)
# 3. 测试功能
# 准备数据
de_genes <- c("TP53", "BRCA1", "EGFR", "BAX", "ATM")
background_genes <- c(de_genes, "AKT1", "MYC", "VEGFA", "PTEN", "CDKN1A")
# 读取GMT文件(或使用自定义基因集)
gene_set <- read_gmt("test_gene_set.gmt")
# 执行富集分析
enrich_result <- custom_enrich(
de_genes = de_genes,
background_genes = background_genes,
gene_set = gene_set,
test_method = "hypergeometric"
)
# 可视化
plot(enrich_result, n_terms = 10, save_path = "enrich_bubble.png", width = 12, height = 8)
# 保存结果
save_enrich_result(enrich_result, "enrich_result.csv")第四章 GitHub 开源规范:从个人开发到社区共享
完成模块封装后,需遵循 GitHub 开源规范,确保代码可被他人理解、复用和贡献。以下是核心规范落地步骤。
4.1 仓库结构设计(通用模板)
plaintext
bioinfo-custom-modules/ # 仓库根目录
├── .github/ # GitHub配置目录
│ ├── ISSUE_TEMPLATE/ # Issue模板(bug报告、功能请求)
│ │ ├── bug_report.md
│ │ └── feature_request.md
│ └── workflows/ # CI/CD工作流(自动测试、部署)
│ └── test.yml # 自动测试配置
├── docs/ # 文档目录(使用Sphinx或mkdocs生成)
│ ├── api/ # API文档
│ ├── tutorials/ # 教程(含案例代码)
│ └── index.md # 文档首页
├── src/ # 源代码目录
│ ├── python/ # Python模块
│ │ └── custom_immune_typing/
│ └── R/ # R包
│ └── CustomEnrichment/
├── tests/ # 测试数据与脚本
│ ├── test_data/ # 测试用数据(如小批量单细胞矩阵)
│ ├── python_test/ # Python测试脚本
│ └── R_test/ # R测试脚本
├── .gitignore # 忽略文件(如__pycache__、.Rhistory)
├── LICENSE # 开源许可证(推荐MIT)
├── README.md # 仓库首页(核心说明)
└── requirements.txt # Python依赖列表(便于他人安装)4.2 核心文件编写
4.2.1 README.md(仓库首页,吸引用户与贡献者)
markdown
# 生信个性化分析模块集合
[](https://github.com/your-username/bioinfo-custom-modules/actions/workflows/test.yml)
[](https://opensource.org/licenses/MIT)
[](https://www.python.org/)
[](https://www.r-project.org/)
## 简介
本仓库包含基于Python和R开发的生信个性化分析模块,解决通用工具无法覆盖的特定研究需求,目前包含:
- **Python模块**:`custom_immune_typing` - 肿瘤单细胞RNA-seq数据的自定义免疫细胞分型
- **R包**:`CustomEnrichment` - 差异表达基因的自定义功能富集分析
所有模块遵循"低耦合、高复用"原则,支持与主流生信工具(Scanpy、DESeq2)无缝集成。
## 快速开始
### 1. 环境准备
#### Python模块
```bash
# 克隆仓库
git clone https://github.com/your-username/bioinfo-custom-modules.git
cd bioinfo-custom-modules
# 安装依赖
pip install -r requirements.txt
# 安装模块(开发模式)
cd src/python
pip install -e .R 包
r
# 安装依赖
if (!require("BiocManager", quietly = TRUE)) install.packages("BiocManager")
BiocManager::install(c("tidyverse", "clusterProfiler", "enrichplot"))
# 安装包
devtools::install_github("your-username/bioinfo-custom-modules", subdir = "src/R/CustomEnrichment")2. 示例代码
Python:单细胞免疫细胞分型
python
import anndata as ad
from custom_immune_typing import (
filter_low_quality_cells,
assign_cell_type,
plot_umap_typing,
save_typing_result
)
# 1. 读取数据(示例数据见tests/test_data/)
adata = ad.read_h5ad("tests/test_data/small_single_cell.h5ad")
# 2. 过滤低质量细胞
adata = filter_low_quality_cells(adata, min_genes=200)
# 3. 自定义细胞分型
adata_typed, stat_df = assign_cell_type(
adata=adata,
marker_gene_path="tests/test_data/immune_marker_genes.csv",
scoring_method="mean",
threshold=0.5
)
# 4. 可视化与结果保存
plot_umap_typing(adata_typed, save_path="./plots")
save_typing_result(stat_df, save_path="./results")R:自定义功能富集分析
r
library(CustomEnrichment)
# 1. 准备数据
de_genes <- readLines("tests/test_data/de_genes.txt")
background_genes <- readLines("tests/test_data/background_genes.txt")
gene_set <- read_gmt("tests/test_data/custom_gene_set.gmt")
# 2. 执行富集分析
enrich_result <- custom_enrich(
de_genes = de_genes,
background_genes = background_genes,
gene_set = gene_set,
test_method = "hypergeometric"
)
# 3. 可视化与结果保存
plot(enrich_result, n_terms = 10, save_path = "./enrich_bubble.png")
save_enrich_result(enrich_result, "./enrich_result.csv")文档
完整文档请查看 在线文档(使用 Sphinx/mkdocs 生成),包含:
模块 API 详细说明完整教程(含真实数据案例)常见问题(FAQ)
贡献指南
欢迎通过以下方式贡献代码:
Fork 本仓库创建特性分支(
git checkout -b feature/new-module
git commit -m "Add new module for X analysis"
git push origin feature/new-module
贡献前请确保:
新增代码包含测试用例文档同步更新遵循代码风格(Python:PEP8;R:tidyverse 风格)
许可证
本项目基于 MIT 许可证 开源,允许自由使用、修改和分发。
联系方式
如有问题,请提交 Issue 或发送邮件至 dev@bioinfo.example.com
plaintext
#### 4.2.2 LICENSE(开源许可证选择)
推荐选择 **MIT许可证**(宽松开源协议,允许他人商用),内容如下:MIT License
Copyright (c) [年份] [作者名]
Permission is hereby granted, free of charge, to any person obtaining a copyof this software and associated documentation files (the “Software”), to dealin the Software without restriction, including without limitation the rightsto use, copy, modify, merge, publish, distribute, sublicense, and/or sellcopies of the Software, subject to the following conditions:
The above copyright notice and this permission notice shall be included in allcopies or substantial portions of the Software.
THE SOFTWARE IS PROVIDED “AS IS”, WITHOUT WARRANTY OF ANY KIND, EXPRESS ORIMPLIED, INCLUDING BUT NOT LIMITED TO THE WARRANTIES OF MERCHANTABILITY,FITNESS FOR A PARTICULAR PURPOSE AND NONINFRINGEMENT. IN NO EVENT SHALL THEAUTHORS OR COPYRIGHT HOLDERS BE LIABLE FOR ANY CLAIM, DAMAGES OR OTHERLIABILITY, WHETHER IN AN ACTION OF CONTRACT, TORT OR OTHERWISE, ARISING FROM,OUT OF OR IN CONNECTION WITH THE SOFTWARE OR THE USE OR OTHER DEALINGS IN THESOFTWARE.
plaintext
#### 4.2.3 .github/workflows/test.yml(CI/CD自动测试)
通过GitHub Actions实现代码推送后自动测试,确保代码质量:
```yaml
name: Test Bioinfo Modules
on:
push:
branches: [ main, develop ] # 推送至这些分支时触发
pull_request:
branches: [ main ] # 提交PR至main分支时触发
jobs:
test-python:
runs-on: ubuntu-latest
steps:
- name: Checkout code
uses: actions/checkout@v4
- name: Set up Python
uses: actions/setup-python@v5
with:
python-version: '3.9'
- name: Install dependencies
run: |
python -m pip install --upgrade pip
pip install -r requirements.txt
pip install -e src/python/
- name: Run Python tests
run: |
cd tests/python_test
pytest -v
test-R:
runs-on: ubuntu-latest
steps:
- name: Checkout code
uses: actions/checkout@v4
- name: Set up R
uses: r-lib/actions/setup-r@v2
with:
r-version: '4.2'
- name: Install R dependencies
run: |
Rscript -e "install.packages('devtools')"
Rscript -e "devtools::install_deps('src/R/CustomEnrichment', dependencies = TRUE)"
- name: Run R tests
run: |
cd tests/R_test
Rscript -e "testthat::test_dir('.')"4.3 版本管理(语义化版本规范)
遵循 语义化版本(Semantic Versioning) 规范,版本号格式为
主版本号.次版本号.修订号
主版本号(Major):不兼容的 API 变更(如函数参数大幅调整);次版本号(Minor):向后兼容的功能新增(如新增可视化函数);修订号(Patch):向后兼容的问题修复(如修复评分计算 bug)。
版本发布步骤:
在
CHANGELOG.md
git tag -a v0.1.0 -m "First release: Python immune typing + R enrichment"
git push origin v0.1.0
第五章 实战进阶:跨语言调用与模块集成
在实际生信分析中,常需结合 Python 和 R 的优势(如用 Python 做单细胞降维,用 R 做差异分析),以下是跨语言调用的实战方案。
5.1 Python 调用 R 模块(基于 rpy2)
在 Python 中直接调用 R 的
CustomEnrichment
python
import rpy2.robjects as robjects
from rpy2.robjects import pandas2ri
from rpy2.robjects.packages import importr
import pandas as pd
# 启用pandas与R数据框的转换
pandas2ri.activate()
# 1. 导入R包(需先安装CustomEnrichment)
custom_enrich = importr("CustomEnrichment")
base = importr("base")
tidyverse = importr("tidyverse")
# 2. 准备Python格式的输入数据
de_genes = ["TP53", "BRCA1", "EGFR", "BAX", "ATM"] # Python列表
background_genes = de_genes + ["AKT1", "MYC", "VEGFA", "PTEN", "CDKN1A"]
# 构造R格式的基因集数据框
gene_set_df = pd.DataFrame({
"term": ["Apoptosis", "Apoptosis", "Apoptosis", "DNA Repair", "DNA Repair", "DNA Repair"],
"gene": ["TP53", "BRCA1", "BAX", "BRCA1", "ATM", "EGFR"]
})
# 转换为R数据框
r_gene_set = pandas2ri.py2rpy(gene_set_df)
# 3. 调用R的custom_enrich函数
enrich_result = custom_enrich.custom_enrich(
de_genes=robjects.StrVector(de_genes),
background_genes=robjects.StrVector(background_genes),
gene_set=r_gene_set,
test_method="hypergeometric",
pvalue_cutoff=0.05,
qvalue_cutoff=0.1
)
# 4. 转换R结果为Python格式
# 提取富集结果(R数据框→Python DataFrame)
enrich_result_df = pandas2ri.rpy2py(enrich_result.rx2("enrichment_result"))
# 提取参数信息(R列表→Python字典)
params = dict(zip(
list(enrich_result.rx2("parameters").names),
[x[0] for x in list(enrich_result.rx2("parameters"))]
))
# 5. 调用R的可视化函数
custom_enrich.plot(enrich_result, n_terms=10, save_path="./r_enrich_bubble.png")
print("富集分析参数:", params)
print("富集结果:")
print(enrich_result_df.head())关键注意事项:
确保 R 环境中已安装
CustomEnrichment
rpy2
robjects.StrVector
robjects.FloatVector
IntVector
5.2 R 调用 Python 模块(基于 reticulate)
在 R 中调用 Python 的
custom_immune_typing
r
# 1. 加载reticulate并配置Python环境
library(reticulate)
# 指定Python环境(与开发时的bio_dev环境一致)
use_condaenv("bio_dev", required = TRUE)
# 2. 导入Python模块
scanpy <- import("scanpy")
custom_immune <- import("custom_immune_typing")
# 3. 读取单细胞数据(R→Python AnnData)
# 方式1:直接读取h5ad文件(Python原生支持)
adata <- scanpy$read_h5ad("tests/test_data/small_single_cell.h5ad")
# 方式2:R数据框→Python AnnData(适用于自定义数据)
# 示例:构造简单的AnnData对象
# expr_matrix <- matrix(rnorm(1000), nrow = 100, ncol = 10) # 100细胞×10基因
# adata <- scanpy$AnnData(X = expr_matrix)
# 4. 调用Python的核心函数
# 过滤低质量细胞
adata_filtered <- custom_immune$filter_low_quality_cells(adata, min_genes=200)
# 自定义免疫细胞分型
marker_gene_path <- "tests/test_data/immune_marker_genes.csv"
typing_result <- custom_immune$assign_cell_type(
adata = adata_filtered,
marker_gene_path = marker_gene_path,
scoring_method = "mean",
threshold = 0.5
)
# 提取结果(Python元组→R列表)
adata_typed <- typing_result[[1]]
stat_df <- py_to_r(typing_result[[2]]) # Python DataFrame→R数据框
# 5. 调用Python的可视化函数
custom_immune$plot_umap_typing(adata_typed, plot_save_path = "./python_umap_typing.png")
# 6. 结果写入R环境分析
cat("细胞分型统计:
")
print(stat_df)
# 提取Python AnnData的注释信息→R数据框
obs_df <- py_to_r(adata_typed$obs)
cat("细胞注释信息:
")
print(head(obs_df))关键注意事项:
reticulate
use_condaenv
use_virtualenv
$
adata$obs
()
scanpy$read_h5ad()
第六章 常见问题与最佳实践
6.1 开发阶段常见问题
| 问题类型 | 典型场景 | 解决方案 |
|---|---|---|
| 依赖冲突 | 安装 R 包时提示 “版本不兼容”、Python 导入模块报错 | 1. 使用虚拟环境隔离依赖;2. 在 DESCRIPTION/requirements.txt 明确版本号;3. 优先用 BiocManager/conda 安装 |
| 跨语言数据转换失败 | rpy2/reticulate 转换数据时抛出异常 | 1. 检查数据类型(如 Pandas 空值→R NA);2. 避免特殊字符列名;3. 小规模数据测试转换逻辑 |
| 函数参数不兼容 | 调用模块时提示 “参数缺失 / 类型错误” | 1. 严格遵循接口文档;2. 为参数设置默认值;3. 新增参数时保持向后兼容 |
| 测试用例覆盖不全 | 上线后发现边界条件 bug | 1. 测试覆盖 “正常输入 / 异常输入 / 空输入 / 大规模输入”;2. 用 pytest/testthat 的参数化测试 |
6.2 开源维护最佳实践
6.2.1 代码风格规范
Python:遵循 PEP8 规范(行宽≤88 字符、变量名小写 + 下划线、函数注释用 Google 风格),用
black
flake8
styler
lintr
6.2.2 文档维护
核心函数必须包含 “输入 / 输出 / 参数说明 / 示例代码”;提供至少 1 个端到端的教程(如基于公开数据集的分析流程);定期更新 FAQ,解答用户高频问题(如 “基因名不匹配如何处理”“低质量细胞过滤阈值怎么选”)。
6.2.3 社区协作
及时响应 Issue(建议 24 小时内回复),标注 Issue 类型(bug/feature/question);对 Pull Request(PR)做代码审查(检查功能完整性、测试覆盖、文档更新);版本发布时明确变更日志(CHANGELOG.md),说明 “新增功能 / 修复 bug / 不兼容变更”。
6.3 性能优化建议
数据读写:大规模单细胞数据优先用 H5AD(Python)/HDF5(R)格式,避免 CSV/TSV(读写慢、占空间);循环优化:Python 中用 NumPy/Scanpy 向量化操作替代 for 循环,R 中用
purrr
dplyr
multiprocessing
furrr
第七章 总结与展望
生信开源工具二次开发的核心是 “基于现有生态,封装个性化逻辑”—— 既无需重复造轮子,又能精准满足特定研究需求。本文通过 Python(单细胞免疫分型)和 R(自定义功能富集)两个实战案例,完整演示了 “需求拆解→模块封装→测试验证→开源发布” 的全流程,同时覆盖跨语言调用、GitHub 开源规范等关键环节。
未来拓展方向
模块化集成:将多个个性化模块整合为完整分析流程(如 “单细胞数据预处理→分型→差异分析→富集分析”),提供一键式运行脚本;可视化增强:结合 Streamlit(Python)/Shiny(R)开发交互式 Web 界面,降低非编程用户的使用门槛;云原生部署:将模块封装为 Docker 镜像,部署到云平台(如 AWS/GCP),支持大规模数据的分布式分析;AI 融合:引入大语言模型(如 GPT-4o、BioBERT)自动生成特征基因列表、解释富集结果,提升分析的智能化水平。
生信分析的核心是 “解决生物学问题”,开源工具二次开发的价值在于让技术服务于科学研究 —— 希望本文的实战方案能帮助生信研究者从 “重复调参” 转向 “定制化分析”,将更多精力投入到生物学意义的解读上。
附录:必备工具与资源清单
开发工具
代码编辑器:VS Code(安装 Python/R 插件)、RStudio;版本控制:Git + GitHub/GitLab;测试工具:pytest(Python)、testthat(R);文档工具:Sphinx(Python)、roxygen2(R)、mkdocs(通用文档);格式检查:black(Python)、styler(R)、flake8(Python)。
学习资源
Python 生信生态:Scanpy 官方文档(https://scanpy.readthedocs.io/)、PyPI 生信包索引(https://pypi.org/search/?q=bioinformatics);R 生信生态:Bioconductor 官方教程(https://bioconductor.org/help/course-materials/)、tidyverse 官方文档(https://www.tidyverse.org/);开源规范:GitHub 开源指南(https://guides.github.com/)、语义化版本规范(https://semver.org/);跨语言调用:rpy2 文档(https://rpy2.github.io/doc/latest/html/)、reticulate 文档(https://rstudio.github.io/reticulate/)。
公开数据集(用于测试)
单细胞 RNA-seq:10x Genomics 公开数据集(https://www.10xgenomics.com/resources/datasets);差异表达基因:TCGA 数据集(https://portal.gdc.cancer.gov/)、GEO 数据集(https://www.ncbi.nlm.nih.gov/geo/);基因集:MSigDB(https://www.gsea-msigdb.org/gsea/msigdb/)、KEGG(https://www.genome.jp/kegg/)。
相关文章